Il a été découvert que les fragments du SRAS-CoV-2 imitent les peptides du système immunitaire et alimentent l’inflammation
[ad_1]
Dans une étude récemment publiée dans la revue Actes de l’Académie nationale des sciencesLes chercheurs ont analysé la capacité inflammatoire des composants fragmentés du coronavirus 2 (SARS-CoV-2) du syndrome respiratoire aigu sévère.
Des recherches intensives pendant la pandémie de maladie à coronavirus 2019 (COVID-19) ont contribué à la compréhension de l’infection par le SRAS-CoV-2. Cependant, on ne sait toujours pas exactement ce qui permet au virus de déclencher une réaction inflammatoire dangereuse. Des études ont montré que les peptides amphiphiles et cationiques du système immunitaire inné forment une connexion de type amyloïde avec les acides nucléiques anioniques et forment des complexes pro-inflammatoires.
 Étude: Vie virale après la mort : le SRAS-CoV-2 comme réservoir de peptides immunomimétiques qui se réassemblent en complexes supramoléculaires proinflammatoires. Crédit image : NIAID
Étude: Vie virale après la mort : le SRAS-CoV-2 comme réservoir de peptides immunomimétiques qui se réassemblent en complexes supramoléculaires proinflammatoires. Crédit image : NIAID
L’étude et les résultats
La présente étude a vérifié si les peptides SARS-CoV-2 fragmentés forment des complexes supramoléculaires avec l’ARN double brin anionique (ARNdb). Le protéome viral était considéré comme un réservoir de fragments peptidiques libérés après destruction protéolytique des virions. Les chercheurs ont utilisé un classificateur de machine à vecteurs de support (SVM) pour détecter les séquences de type peptide antimicrobien (AMP) (XenoAMP) dans le protéome du SRAS-CoV-2.
Les séquences de protéines virales ont été analysées sur une fenêtre mobile de 24 à 34 acides aminés pour identifier les xénoAMP potentiels et vérifier s’ils se comportent comme des AMP lorsqu’ils sont clivés à différentes positions. Les séquences ont été sélectionnées sur la base du résultat fourni par le classificateur sous la forme d’un score sigma (σ), avec un score fortement positif impliquant que la séquence était très probablement un AMP.
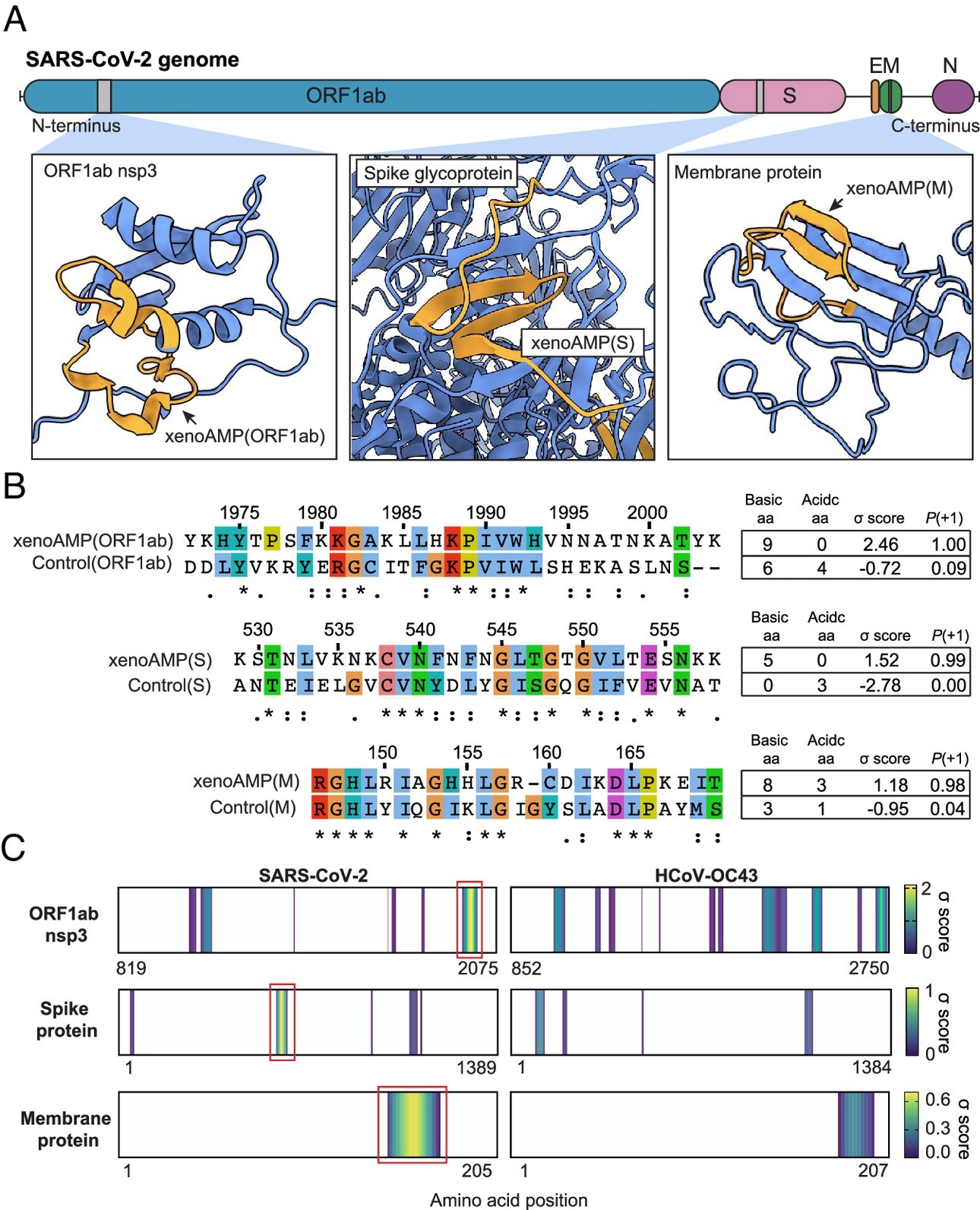
Présence d’imitations exogènes de peptides hôtes antimicrobiens proinflammatoires (XenoAMP) dans les protéines du SRAS-CoV-2. (A) Les protéines du SRAS-CoV-2 sont analysées avec un classificateur AMP d’apprentissage automatique. Chaque séquence interrogée reçoit un score σ qui mesure sa qualité AMP. Trois séquences représentatives à score élevé sont examinées : xenoAMP(ORF1ab), xenoAMP(S) et xenoAMP(M). Les barres grises marquent l’emplacement où les séquences correspondantes sont sélectionnées. (B) Les séquences du SRAS-CoV-2 sont alignées et comparées à leurs homologues dans un coronavirus du rhume HCoV-OC43 : contrôle (ORF1ab), contrôle (S) et contrôle (M). Les astérisques, les deux-points et les points indiquent respectivement les positions avec des résidus entièrement conservés, ceux avec des propriétés fortement similaires et ceux avec des propriétés faiblement similaires. Chaque résidu se voit attribuer une couleur à l’aide du schéma ClustalX. (C) Les cartes thermiques σ-score comparent la distribution des séquences à score élevé dans trois protéines du SRAS-CoV-2 et du HCoV-OC43. Le premier acide aminé de chaque séquence est coloré en fonction de son score σ moyen ; Les régions avec des valeurs σ moyennes négatives (non-AMP) sont colorées en blanc. Les groupes de « points chauds » de séquences à score élevé pour le SRAS-CoV-2 (régions jaune clair dans les cases rouges) présentent des scores systématiquement plus élevés et couvrent de plus grandes régions de l’espace de séquence par rapport au HCoV-OC43. Cette tendance suggère que les points chauds du SRAS-CoV-2 pourraient générer des séquences de notation plus élevées pour une plus grande variété de sites de clivage enzymatique que celles du HCoV-OC43.
De plus, l’équipe a sélectionné des séquences spécifiques parmi cette population de séquences (très bien notées) à forte charge cationique. Plus précisément, ils se sont concentrés sur des candidats prototypiques de la protéine membranaire (M), de la protéine de pointe (S) et de la polyprotéine à cadre de lecture ouvert 1ab (ORF1ab). In silico Les analyses ont montré que ces xénoAMP peuvent être générés lors de la dégradation protéasomale, avec la métalloprotéinase matricielle 9 (MMP9) et l’élastase neutrophile (NE) capables de les générer.
Ensuite, l’équipe a comparé les xenoAMP du SRAS-CoV-2 avec des séquences homologues du SRAS-CoV-1 et des CoV humains non pandémiques. Cela a montré que les séquences étaient partiellement conservées. Une comparaison des cartes thermiques à score σ des protéines ORF1ab, S et M entre le SRAS-CoV-2 et le HCoV-OC43 a révélé que les séquences à score élevé étaient regroupées en points chauds, les points chauds du SRAS-CoV-2 ayant des scores plus élevés et répartis sur des points chauds plus grands. régions que celle du HCoV-OC43.
De plus, la spectrométrie de masse a été réalisée sur des échantillons d’aspiration trachéale provenant de patients atteints d’une forme grave de la maladie COVID-19. L’équipe a découvert des fragments de l’AMP hôte, la cathélicidine LL-37, dans 20 échantillons (sur 29). En revanche, 28 échantillons contenaient des fragments de peptides viraux, dont certains avaient des valeurs σ suffisamment élevées pour être considérés comme des xénoAMP.
Les trois xenoAMP, XenoAMP(S), XenoAMP(M) et Polyinosinique : l’acide polycytidylique (Poly(I:C)) a été utilisé comme analogue synthétique pour imiter l’ARNdb viral produit lors de la réplication. Les structures des complexes xénoAMPs-poly(I:C) étaient liées aux complexes hôtes AMPs-ARNdb.
Ensuite, l’équipe a examiné la robustesse de ces complexes pro-inflammatoires auto-assemblés dans des conditions sous-optimales. Ils ont constaté que les structures nanocristallines étaient préservées lorsque les XenoAMP impliqués étaient raccourcis. De plus, les xénoAMP du SRAS-CoV-2 se sont co-cristallisés avec le LL-37, ce qui suggère que les AMP de l’hôte et les xénoAMP pourraient activer de manière synergique les réponses inflammatoires.
La capacité d’activation immunitaire des xénoAMP du SARS-CoV-2 a été comparée à celle des peptides homologues du HCoV-OC43 à l’aide de monocytes humains. Les monocytes traités au XenoAMP-Poly(I:C) ont libéré 1,7 fois plus d’interleukine (IL)-8 que les témoins traités au poly(I:C). En revanche, les complexes formés avec des peptides homologues du HCoV-OC43 ont induit des taux d’IL-8 beaucoup plus faibles.
De plus, la stimulation par xenoAMP-poly(I:C) des cellules endothéliales microvasculaires dermiques humaines primaires (HDMVEC) a déclenché une forte production d’IL-6, qui n’a pas été observée avec les complexes peptidiques HCoV-OC43. Notamment, les HDMVEC traitées avec xenoAMP-poly (I: C) ont montré une régulation positive significative de plusieurs gènes de chimiokines et de cytokines proinflammatoires.
Enfin, les chercheurs ont mesuré la capacité d’activation immunitaire chez la souris. Les souris C57BL/6 non exposées à l’infection ont été traitées avec des complexes xenoAMP(ORF1ab)-poly(I:C) ou poly(I:C) seul (témoin). Traitement avec 2,2 fois. De plus, les niveaux d’IL-6 et de CXCL1 dans les poumons ont augmenté de 1,2 fois par rapport au traitement témoin.
Conclusions
En résumé, l’étude a révélé un mécanisme inattendu de propagation inflammatoire à travers des cellules non infectées dans le COVID-19, dans lequel des fragments de virus imitent les AMP tels que le LL-37. Cela pourrait être important pour comprendre pourquoi le système immunitaire de l’hôte dans le cas du COVID-19 est similaire à celui des personnes atteintes de maladies auto-immunes telles que la polyarthrite rhumatoïde et le lupus.
Les chercheurs ont découvert que les protéases de l’hôte peuvent générer des xenoAMP, ce qui suggère que les inhibiteurs de protéase qui suppriment la génération de xenoAMP pourraient avoir un impact clinique sur l’inflammation induite par le virus. La dégradation protéolytique du SRAS-CoV-2 pourrait différer d’un hôte à l’autre, expliquant potentiellement l’hétérogénéité des résultats de l’infection, par ex. B. asymptomatique et mortel.
Référence du magazine :
- Zhang Y, Bharathi V, Dokoshi T et al. La vie virale après la mort : le SRAS-CoV-2 comme réservoir de peptides immunomimétiques qui se réassemblent en complexes supramoléculaires proinflammatoires. Proc Natl Acad Sci États-Unis2024, EST CE QUE JE: 10.1073/pnas.2300644120, https://www.pnas.org/doi/10.1073/pnas.2300644120
[ad_2]
Source
