Dans une étude récemment publiée dans la revue Nature, Les chercheurs ont mené des recherches génomiques de pointe sur des restes humains brésiliens vieux de 2 000 ans pour étudier l’évolution et les origines des génomes tréponémiques ancestraux dans les Amériques et dans l’Ancien Monde. Ils reconstruisent quatre de ces génomes anciens et découvrent qu’ils sont les plus proches parents existants du pathogène tréponémique préhistorique. Treponema pallidum endémique, l’agent pathogène responsable de Bejel. Ces résultats remettent en question les hypothèses antérieures concernant la caractérisation des sous-types de tréponèmes et soulignent que les tréponèmes sont beaucoup plus adaptables qu’on ne le pensait auparavant.
 Étude: Redéfinir l’histoire des tréponèmes à l’aide des génomes précolombiens du Brésil. Source de l’image : bekirevren / Shutterstock
Étude: Redéfinir l’histoire des tréponèmes à l’aide des génomes précolombiens du Brésil. Source de l’image : bekirevren / Shutterstock
Que sont les tréponèmes ?
Les tréponèmes sont un genre de bactéries en forme de spirale, dont certaines sont responsables d’infections tréponémiques telles que la syphilis. Tréponème pallidum pallidum (TPA)), Béjel (T. pallidum endémique (DIX)) et le lacet (T. pallidum pertenue (TPE)). Alors que les antibiotiques conventionnels ont considérablement réduit le fardeau de ces agents pathogènes, des recherches récentes révèlent de plus en plus de preuves que les tréponèmes développent une résistance à plusieurs antibiotiques, conduisant à une relance des études médicales et scientifiques sur ces bactéries.
Bien que TPA, TEN et TPE ne varient que de 0,03 % dans leurs séquences génomiques, ils ont des écologies et des pathologies remarquablement différentes. Le Bejel se trouve dans les régions chaudes et sèches de l’Asie occidentale et de la Méditerranée orientale, l’Aria est limitée aux régions tropicales humides de l’Afrique et de l’Amérique du Sud, et la syphilis est mondiale. Les dossiers médicaux indiquent que même si la syphilis survient quel que soit le statut de développement régional de ses hôtes, le béjel et le pian ne surviennent que dans les pays en développement et ont été éradiqués dans les pays développés.
Que sait-on de leur histoire ?
la syphilis et T. pallidum pallidum ont suscité beaucoup plus d’intérêt académique que Bejel et Pian, même si les trois maladies présentent des symptômes et des interventions cliniques similaires. Cela est probablement dû à la notoriété de la première : la syphilis a été responsable d’une épidémie dévastatrice en Europe en 1495. Bien que l’Europe ait été suggérée comme lieu d’origine potentiel des souches tréponémiques, il existe encore une abondante littérature testant cette hypothèse, qui est compliquée par les diagnostics de syphilis d’infections historiques, qui laissent des traces visibles dans seulement 5 à 30 % des cas avancés.
Il existe actuellement deux hypothèses concurrentes concernant l’apparition de la syphilis en Europe : l’hypothèse précolombienne et l’hypothèse colombienne. La première est étayée par des preuves paléopathologiques et suppose que la bactérie existait en Europe des centaines ou des milliers d’années avant la première expédition américaine de Colomb et qu’elle y était probablement originaire. En revanche, la seconde suppose que ce sont Colomb et les colons ultérieurs qui ont introduit les tréponèmes sur le continent.
Les quelques études qui ont tenté de déchiffrer la génétique des tréponèmes se sont révélées difficiles étant donné le manque criant d’ADN de tréponème ancien. Comprendre l’histoire évolutive et la systématique de ces agents pathogènes éclairerait les études futures sur les stratégies optimales pour se préparer à d’éventuelles épidémies.
À propos de l’étude
Dans la présente étude, les chercheurs ont utilisé des restes humains vieux de près de 2 000 ans provenant du lieu de sépulture de Jabuticabeira II pour reconstruire quatre ancêtres. T. pallidum Génomes avec une couverture jusqu’à 33,6x. Le groupe d’échantillon comprenait 96 spécimens du lieu de sépulture, avec (n = 32) et sans suspicion de pathologies tréponémiques. Un examen préliminaire a identifié 37 échantillons contenant de l’ADN tréponémique utilisable.
Tous les échantillons ont été datés au radiocarbone, quatre échantillons d’os fournissant l’ADN génomique requis. Le séquençage au fusil de chasse a été utilisé pour le dépistage préliminaire des agents pathogènes. L’enrichissement cible de l’ADN séquencé a donné neuf échantillons avec plus de 5 000 mappages de lecture T. pallidum Génomes de référence (achetés auprès de BosniaA, CDC2 et Nichols). Les quatre échantillons d’os mentionnés ci-dessus (ZH1390, ZH1540, ZH1541 et ZH1557) se sont avérés avoir des lectures couvrant 9,2 à 99,4 % du génome de référence de Bosnie-Herzégovine.
La désamination de la base a été estimée pour vérifier l’authenticité de l’ADN ancien récupéré. Le séquençage Illumina à haut débit de ZH1390, ZH1540, ZH1541 et ZH1557 a été utilisé pour générer des lectures pour la reconstruction du génome ancestral. Ces lectures ont ensuite été cartographiées sur les génomes de référence TPE (CDC2), TEN (BosniaA) et TPA (Nichols).
“La séquence finale obtenue pour l’échantillon ZH1540 a donné une couverture de 99,38 % par rapport au génome de référence TEN (BosniaA), une profondeur de couverture minimale de 5× et une profondeur moyenne de 33,6×.”
L’ancien génome ZH1540 nouvellement généré a été assemblé (aligné) et analysé avec 98 génomes de tréponèmes, dont TEN (8 souches), TPE (30 souches) et TPA (60 souches), tous issus du National Center for Biotechnology Information (NCBI). . ont été acquis ) et les bases de données de l’European Nucleotide Archive (ENA). L’alignement résultant était de 1 141 812 nucléotides et comprenait 6 149 polymorphismes nucléotidiques simples (SNP).
Les modèles d’héritage et les analyses phylogénétiques ont été réalisés à l’aide d’une analyse de recombinaison avec la méthode d’incongruence phylogénétique (PIM). La relation a été visualisée à l’aide d’approches de construction d’arbres à vraisemblance maximale. Pour déterminer l’âge du génome reconstruit, une datation par horloge moléculaire a été réalisée.
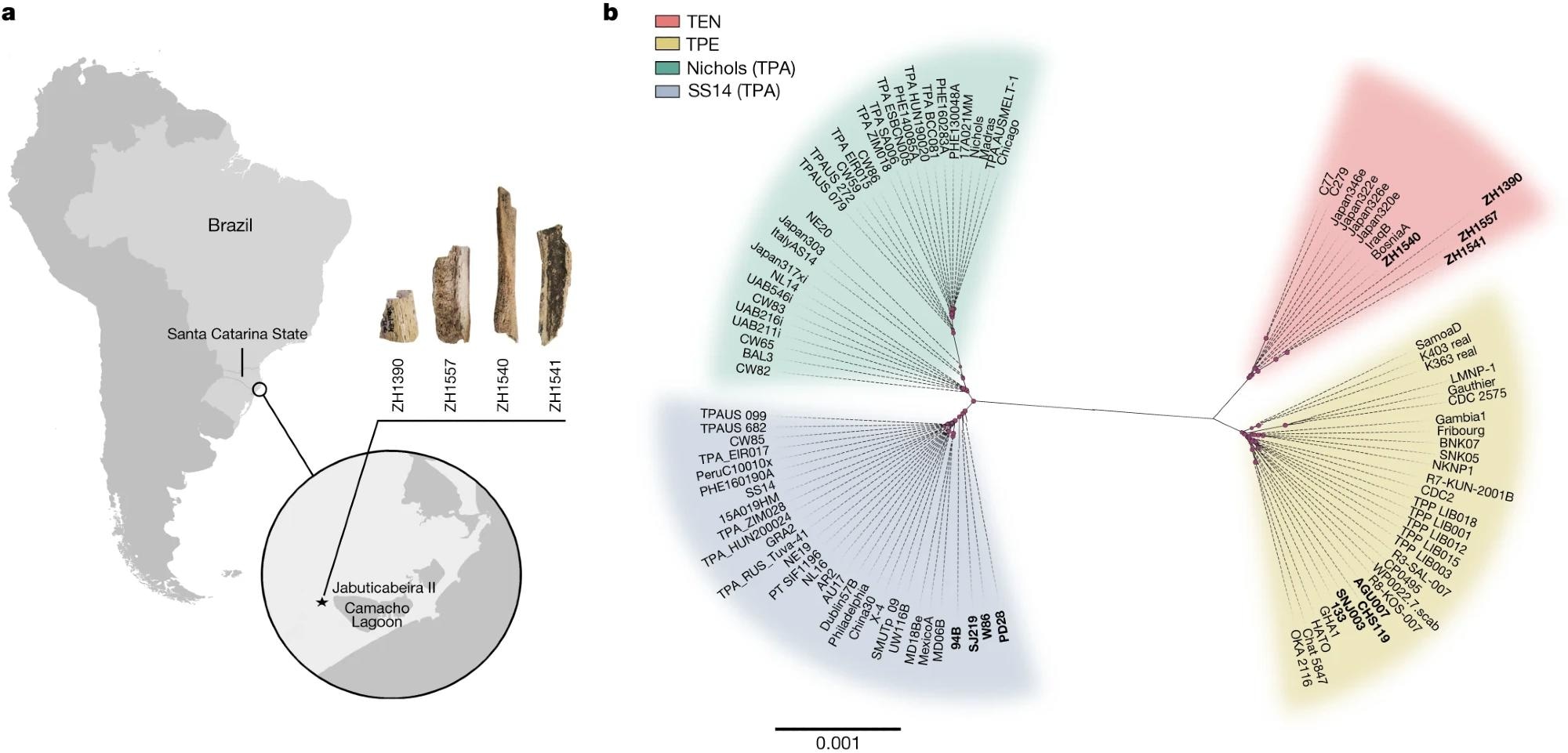 UNUne carte montrant l’emplacement du site archéologique de Jabuticabeira II sur la côte sud de l’État brésilien de Santa Catarina et des échantillons ZH1390, ZH1540, ZH1541 et ZH1557, dont les génomes ont été reconstruits. b, Un arbre phylogénétique à vraisemblance maximale de souches modernes et anciennes de T. pallidum utilisant GTR + G + I comme modèle évolutif et 1 000 répétitions bootstrap. Tous les génomes anciens utilisés dans cette étude (génomes anciens récemment reconstruits et publiés précédemment ; voir le tableau supplémentaire). 3) sont marqués en gras. Les points roses représentent les nœuds avec des valeurs de bootstrap supérieures à 70 %. La barre d’échelle est en unités par remplacement et par site.
UNUne carte montrant l’emplacement du site archéologique de Jabuticabeira II sur la côte sud de l’État brésilien de Santa Catarina et des échantillons ZH1390, ZH1540, ZH1541 et ZH1557, dont les génomes ont été reconstruits. b, Un arbre phylogénétique à vraisemblance maximale de souches modernes et anciennes de T. pallidum utilisant GTR + G + I comme modèle évolutif et 1 000 répétitions bootstrap. Tous les génomes anciens utilisés dans cette étude (génomes anciens récemment reconstruits et publiés précédemment ; voir le tableau supplémentaire). 3) sont marqués en gras. Les points roses représentent les nœuds avec des valeurs de bootstrap supérieures à 70 %. La barre d’échelle est en unités par remplacement et par site.
Résultats de l’étude
La présente étude a généré quatre génomes tréponémiques anciens, qui étaient tous, de manière inattendue, les plus étroitement liés à l’agent causal Bejel. T. pallidum endémique. Bien que la syphilis et le pian aient été identifiés à partir d’anciens génomes de l’Ancien et du Nouveau Monde, ce sont les premiers agents pathogènes de type TEN jamais isolés à partir de vestiges archéologiques.
“Nos résultats dans cette étude ne font que confirmer ce point de vue : un ancien pathogène de type TEN identifié dans une région côtière humide du Brésil, loin de la niche géographique actuelle de la maladie, témoigne de la capacité des tréponèmes à s’adapter à différentes zones climatiques et géographiques. » . L’ADN tréponémique de haute qualité récupéré d’une source préhistorique confirme l’utilisation d’anciennes techniques d’ADN pour former une hypothèse entièrement nouvelle et plus éclairée sur les événements qui ont conduit à la propagation mondiale de Treponema pallidum.
Les analyses de l’horloge moléculaire montrent que T. pallidum La cohorte est beaucoup plus ancienne qu’on ne le pensait, de plus de 1 000 ans. La cohorte est également considérée comme très adaptable, ce qui suggère que leur ascendance s’étend bien au-delà de celle d’aujourd’hui.
« Parce que la découverte révolutionnaire de la tréponématose précolombienne est le résultat d’une combinaison de génomique d’agents pathogènes anciens et d’une sélection minutieuse d’échantillons archéologiques, nous pouvons nous attendre à ce que les découvertes futures mettent en lumière les événements qui ont conduit à l’essor et à la propagation des maladies vénériennes. syphilis « et aider à élucider les facteurs évolutifs responsables du succès mondial de la famille Treponema ».
Référence du magazine :
- Majander, K., Du Plessis, L., Arora, N., Filippini, J., Eggers, S. et Schuenemann, VJ (2024). Redéfinir l’histoire des tréponèmes à l’aide des génomes précolombiens du Brésil. Nature1-7, DOI – 10.1038/s41586-023-06965-x, https://www.nature.com/articles/s41586-023-06965-x
